海水に含まれるDNAから外洋の小型浮魚類の分布を探る
2022年9月8日
東京大学 大気海洋研究所
研究成果
発表のポイント
◆魚類の分布密度が低い外洋域において、海水に含まれるDNAからマイワシなどの小型浮魚の分布を検出可能なことを示しました。
◆小型浮魚に限定したDNA量を定量分析するqPCR法と、全魚種のDNAを同時に分析するメタバーコーディング法の両者で、類似した小型浮魚の分布が検出されることを検証しました。
◆両手法を組み合わせて利用することで、小型浮魚の定量的な分布に加え、小型浮魚を取り巻く多様な魚類群集を推定することができ、生態系に準拠した資源管理に貢献することが期待されます。
発表者
余 泽庶(東京大学大学院農学生命科学研究科水圏生物科学専攻 博士課程)
伊藤 進一(東京大学大気海洋研究所 教授)
由上 龍嗣(水産・研究教育機構水産資源研究所 グループ長)
兵藤 晋(東京大学大気海洋研究所 教授)
発表概要
魚類から海水中に放出されたDNA(環境DNA)を分析することによって、魚類の分布を調べる方法が沿岸域で利用されるようになってきましたが、外洋域での応用(OceanDNA)は、海水中に含まれる環境DNA濃度が低く、実用化に疑問が残されていました。
そこで、東京大学大気海洋研究所の伊藤進一教授、兵藤晋教授、水産・研究教育機構水産資源研究所の由上龍嗣グループ長らを中心とする研究チームは、自ら開発したマイワシ、カタクチイワシ、マサバ、ゴマサバ、マアジ、サンマの6種の小型浮魚類のDNA量を定量分析するqPCR法(注1)(Wong et al., 2022)と、全魚種を同時に分析するメタバーコーディング法(注2)を利用して、黒潮続流域(注3)で収集した海水サンプルから小型浮魚類(注4)の分布を調査しました。両手法での検出結果が類似していることが確認され、OceanDNAの有効性が確認されました。qPCR法は定量的な情報も得られますが、対象魚種6種だけの情報しか得られません。メタバーコーディング法による全魚種の相対的な分布情報と組み合わせることで、小型浮魚類を取り巻く魚類魚群集構造を同時に推定する方法を提案しました。
本研究成果は、2022年9月7日(米国東部夏時間)に米国科学誌「PLOS ONE」のオンライン版に掲載されました。
発表内容
<研究の背景と問題点>
海はその広大さに加え、変化しやすい天候のため、広域を回遊する魚類の分布を把握することは困難です。一方、湖や河川あるいは沿岸域においては、水生動物から放出されたDNA(鱗や粘液など様々な形で放出)である環境DNAを分析することによって、魚類をはじめとする水生生物の分布を調べる方法が多く利用されるようになってきました。しかし、外洋域では、魚類の分布密度が低いため、海水中に含まれる環境DNA濃度が低く、外洋域での環境DNAよる魚類分布把握(OceanDNA)が可能か不明な点が残されていました。
環境DNAの分析手法は、種特異的解析(定量的ポリメラーゼ連鎖反応(quantitative polymerase chain reaction: qPCR)と網羅的解析(メタバーコーディング法)に大別されます。qPCR法は、分析対象とする魚種の分布量を定量的に把握することが可能ですが、一度に解析できる魚種が限定されます。一方、メタバーコーディング法は、全魚種を対象に同時並行的に分析でき、海水サンプル中に含まれるDNAの相対的存在比を求めることが可能ですが、サンプル間の定量的比較には問題点も残されています。
<研究内容>
この研究では、qPCR法とメタバーコーディング法を用いて外洋域で採取した海水サンプルを分析することで、OceanDNAの有効性を試験しました。対象としては、水産物として重要であり、かつ広範囲を回遊する小型浮魚類であるマイワシ、カタクチイワシ、マサバ、ゴマサバ、マアジ、サンマの6魚種を選択しました。これらの魚種は、水温特性が異なるため、水温前線を跨いで分布が変化することが予想されます。このため、2018年5月に東北海洋生態系調査研究船(学術研究船)新青丸にて、顕著な水温前線が形成される黒潮続流を横断するような観測線(図1)で、水深0mから300mまでの海水を採取し、qPCR法とメタバーコーディング法で分析しました。ただし、今回用いたメタバーコーディング法では、マサバとゴマサバのDNA配列が類似しているため両種を区別できないため、マサバとゴマサバをサバ類としてまとめて、4魚種1魚類の分布を調べました。また、同時期に同海域で、水産研究・教育機構の漁業調査船蒼鷹丸が表層トロール網を用いて採取した漁獲物の組成との比較も実施しました。
表層トロールの漁獲、表層トロールに最も近い観測点でのqPCR法およびメタバーコーディング法による検出のいずれにおいてもマイワシ、カタクチイワシ、サバ類が含まれており、マイワシが優占していました。また、マアジとサンマは含まれていませんでした(図2)。これらの結果からOceanDNAの有効性が確認されました。
qPCR法とメタバーコーディング法による小型浮魚類の検出を全観測点で合計すると、両者ともマイワシ、サバ類、カタクチイワシ、サンマ、マアジの順に検出率が高く、傾向が一致していました。また、両者の検出傾向に統計的にも相関があることが示され、検出された小型浮魚類の分布も類似していました(図3)。これらの結果は、qPCR法とメタバーコーディング法ともに外洋域においても小型浮魚類の検出に有効であることを示しています。
観測域の広い範囲で分布していたマイワシ、サバ類、カタクチイワシがqPCR法で検出されたサンプルのメタバーコーディング法結果から、マイワシ、サバ類、カタクチイワシを取り巻く魚類魚群集構造を推定しました(図4)。このようにqPCR法から対象魚種6種の定量的な情報を得ることができ、同時にメタバーコーディング法から対象魚種を取り巻く魚類魚群群集耕造を推定する方法を提案しました。
<社会的意義と今後の展望>
本研究によりOceanDNAによる小型浮魚類の分布モニタリングが可能なことが示されました。OceanDNAは魚類を捕殺することなく分布を調査することが可能です。また、OceanDNAを用いることで、漁網を用いた調査では不可能だった時空間的に高解像度な観測が可能となります。従って、OceanDNAを用いることで、海洋前線や渦などの顕著な海洋構造に対応した小型浮魚類の分布を把握したり、短期間での小型浮魚類の分布変化を把握したりすることが可能になると期待されます。
さらに、本研究で提案した手法を用いることで、小型浮魚類の分布だけでなく、小型浮魚類を取り巻く魚類群集構造を同時に推定することが可能となり、海洋生態系に準拠した資源管理が進展することが期待されます。
本研究は、科研費「サンマ初期生活史の回遊経路の非連続性と分布沖合化維持機構の解明(課題番号:JP21H04735)」、「東京大学FSIプロジェクト(オーシャンDNA:海洋DNAアーカイブ・解析拠点による太平洋の生物多様性と生物資源の保全」の支援により実施されました。この論文は水産庁水産資源調査・評価推進委託事業による成果の一部を活用しています。
関連論文:
Wong M. K., S. Nobata, S. Ito and S. Hyodo (2022) Development of species-specific multiplex real time PCR assays for tracing the small pelagic fishes of North Pacific with environmental DNA. Environmental DNA, 4, 510-522. http://doi.org/10.1002/edn3.275![]()
発表雑誌
雑誌名:「PLOS ONE」(9月7日付)
論文タイトル:Comparison of species-specific qPCR and metabarcoding methods to detect small pelagic fish distribution from open ocean environmental DNA
著者:Zeshu Yu, Shin-ichi Ito*, Marty Kwok-Shing Wong, Susumu Yoshizawa, Jun Inoue, Sachihiko Itoh, Ryuji Yukami, Kazuo Ishikawa, Chenying Guo, Minoru Ijichi, Susumu Hyodo
DOI番号:10.1371/journal.pone.0273670
アブストラクトURL:https://journals.plos.org/plosone/article?id=10.1371/journal.pone.0273670![]()
問い合わせ先
東京大学大気海洋研究所 海洋生命システム研究系 海洋生物資源部門
教授 伊藤 進一(いとう しんいち)
E-mail:goito◎aori.u-tokyo.ac.jp ※「◎」は「@」に変換してください
用語解説
- 注1:qPCR法
- 特定の種のDNAを検出するために、ポリメラーゼ連鎖反応(polymerase chain reaction: PCR)を用いて解析対象遺伝子領域を増幅させる方法のうち、定量的にDNA量を求める方法をqPCR(quantitative PCR)法と呼ぶ。本研究では、Wong et al. (2022)で開発したマイワシ、カタクチイワシ、マサバ、ゴマサバ、マアジ、サンマの6種の小型浮魚類のDNAを検出する方法を用いた。
- 注2:メタバーコーディング法
- 種間でDNAに変化があるDNA領域データベースと照合することで、全魚種を網羅的に検出する方法。本研究では、国内で開発されたMiFishユニバーサルPCRプライマーセットを用いた。
- 注3:黒潮続流
- 本州南岸を流れる海流である黒潮は、房総半島沖で日本列島から離岸し、東向きに向きを変える。この東向きの流れを黒潮続流と呼ぶ。
- 注4:小型浮魚
- マイワシ、カタクチイワシ、マサバ、ゴマサバ、マアジ、サンマなど、海洋表層に分布する小型魚の総称。
添付資料
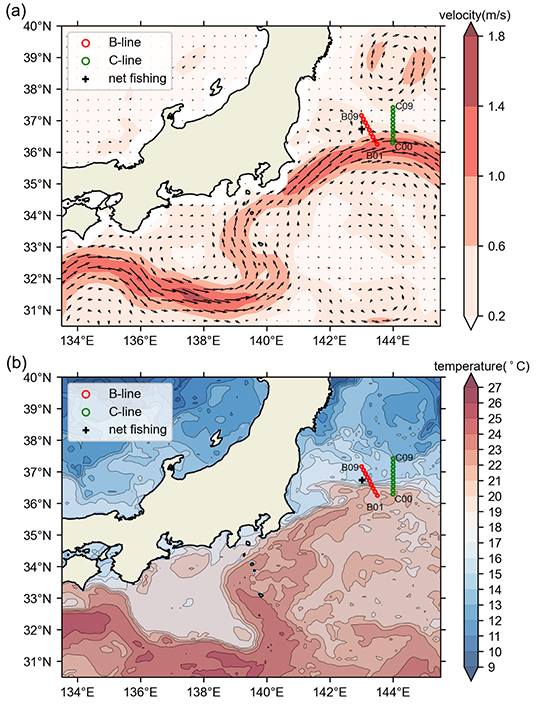
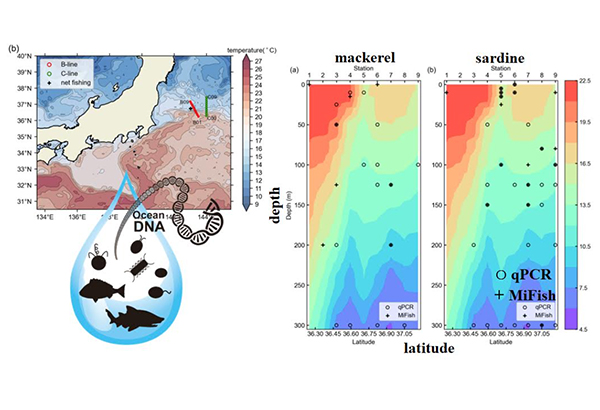
図1 . 本研究のOceanDNAサンプル採集観測点の分布(赤丸および緑丸)および比較に用いた表層トロール実施観測点(+印)。背景は、観測期間中の(a)色が流速を矢印が流向および流速、(b)色が海面水温、を表す。(a)はOcean Surface Current Analysis Real-time data (https://sealevel.jpl.nasa.gov/documents/1641/![]() ), (b)はThe contours and colors represent sea surface temperature (˚C) distribution on May 10, 2018, from GHRSST Level 4 OSTIA Global Foundation Sea Surface Temperature Analysis data (https://podaac.jpl.nasa.gov/dataset/UKMO-L4HRfnd-GLOB-OSTIA
), (b)はThe contours and colors represent sea surface temperature (˚C) distribution on May 10, 2018, from GHRSST Level 4 OSTIA Global Foundation Sea Surface Temperature Analysis data (https://podaac.jpl.nasa.gov/dataset/UKMO-L4HRfnd-GLOB-OSTIA![]() )よりデータを取得。
)よりデータを取得。
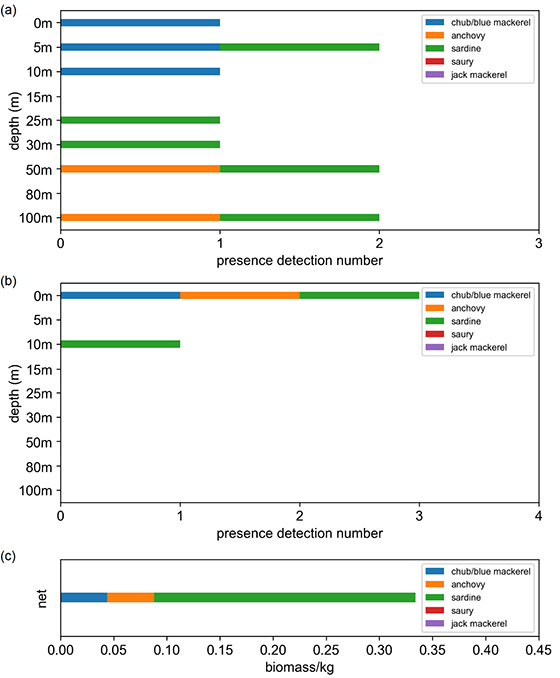
図2. 表層トロール観測点から最も近い観測点B06において検出されて小型浮魚類((a) qPCR法、(b) メタバーコーディング法)と(c) 表層トロールにおける小型浮魚類の漁獲量の比較。
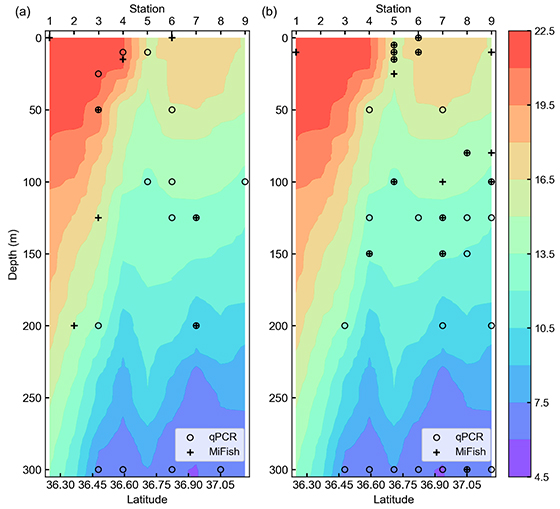
図3. 観測線B鉛直断面においてOceanDNAから検出された(a)サバ類、(b)マイワシの分布(○:qPCR法、+:メタバーコーディング法)。
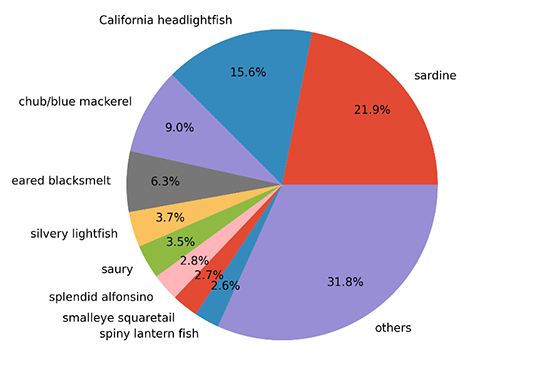
図4.qPCR法によってマイワシ、サバ類、カタクチイワシが検出されたサンプルを用いてメタバーコーディング法を実施した際に検出された魚種のDNA検出量比
sardine: マイワシ、California headlightfish: トドハダカ、chub/blue mackerel: マサバおよびゴマサバ、eared blacksmelt: ソコイワシ、silvery lightfish: キュウリエソ、saury: サンマ、splendid alfonsino: キンメダイ、smalleye squaretail: ドクウロコイボダイ、spiny lantern fish: イバラハダカ。
![]()

 教職員募集
教職員募集 所内専用
所内専用
