つながるサクラエビ ―日本・台湾間における遺伝的交流を解明―
2025年3月5日
東京大学
東海大学

研究成果
発表のポイント
◆近年、漁獲量の減少が続く駿河湾の宝石“サクラエビ”ですが、その生態に関わる科学的知見は圧倒的に不足しています。
◆漁獲対象となっている駿河湾および台湾のサクラエビで複合的な集団遺伝解析を行ったところ、日本-台湾間のサクラエビは遺伝的に交流していることが明らかになりました。
◆本研究の結果はサクラエビの海を跨いだ「つながり」を示すものであり、今後の適切な資源管理や生態理解には日本と台湾の相互協力が不可欠であると考えられます。

図1:駿河湾の宝石“サクラエビ”
発表内容
東京大学大気海洋研究所の平井惇也講師と、東海大学海洋学部海洋生物学科の西川淳教授らによる研究グループは、駿河湾および台湾島北部・南西部のサクラエビを複合的な分子生物学的手法により解析し、日本-台湾間における遺伝的交流を調査しました。
サクラエビは成体で4~5cmまで成長するエビの一種で、日本では主に静岡県の駿河湾で漁獲され、その高い栄養価や見た目の美しさから「駿河湾の宝石」と称されています(図1)。一方、駿河湾においては2010年代後半からサクラエビの記録的不漁が続き、その持続的な利用が危ぶまれています。しかし、サクラエビは科学的知見に基づいた調査が不足しており、その生態の多くは未だに謎に包まれています。例えば、サクラエビは駿河湾の他に台湾北東部および南東部でも漁獲され、台湾産のサクラエビは日本にも輸出されています。これらの日本産および台湾産のサクラエビは異なる集団として捉えられていますが、最新の分子生物学的手法による研究は行われておらず、資源管理の上でも重要な日本-台湾間の遺伝的交流の詳細は不明でした。
そこで本研究グループは台湾農業部水産試驗所の研究者と共同研究を行い、最新の分子生物学的手法を含めた複合的な解析により日本-台湾間におけるサクラエビの遺伝的交流の実態を調べました。初めに、従来法であるミトコンドリアDNA(注1)の特定領域(約500塩基)を用いた解析を行ったところ、従来法ではサクラエビの集団構造を正確に捉えることが困難なことが確かめられました。次に、ゲノム上の一塩基多型(注2)を効率的に解析することが可能なMIG-seq法(注3)を用いた集団遺伝解析を行ったところ、駿河湾および台湾のサクラエビで明確なグループが形成されず、日本-台湾間に遺伝的交流を示す結果が得られました(図2)。また、一塩基多型のデータをもとに現在の移動度を推定したところ、台湾から日本への集団の移動が大きいことが分かり、流れの速い黒潮がサクラエビの分散にも大きく関わっていると考えられました(図2)。さらに、ゲノムスキミング(注4)法によってサクラエビのミトコンドリアゲノムの全長(約16000塩基)を取得したところ、こちらも駿河湾と台湾で明確な遺伝的グループが形成されず、MIG-seq法の結果を支持する結果となりました。
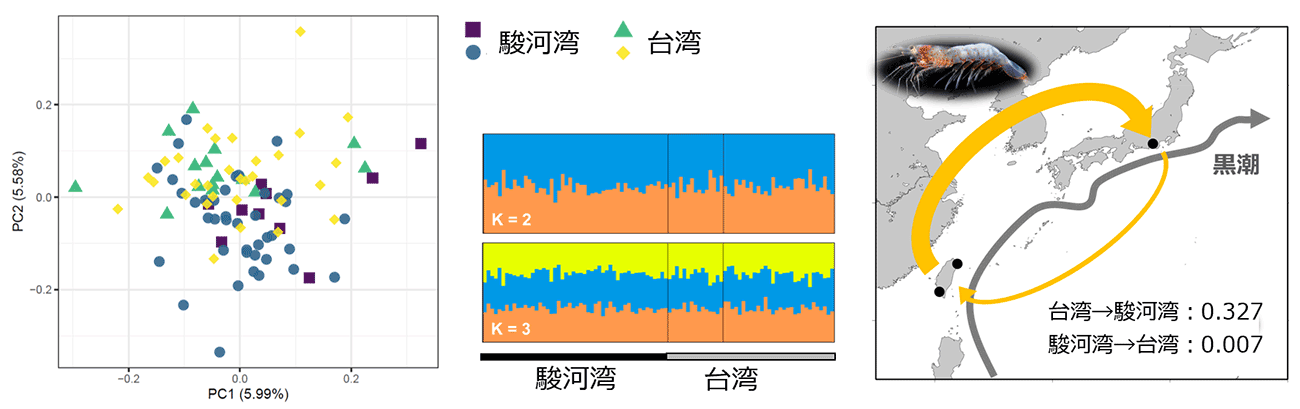
図2:MIG-seq法によるサクラエビの集団遺伝解析
(左)一塩基多型に基づく主成分分析。駿河湾と台湾の個体が混ざり合い、各地点で明確なグループが形成されていない。(中央)一塩基多型に基づくクラスタリング解析。特定の集団数(K)を想定した際に各個体がどの集団に帰属するかが色で表される。駿河湾および台湾で明確に色が分かれておらず、明確な集団構造が形成されていない。(右)一塩基多型から推定された相対的な移動度。
本研究は最新の複合的な分子生物学的手法を駆使し、台湾との国際共同研究によりサクラエビの日本-台湾間の遺伝的交流を明らかにした初の研究となります。サクラエビはどのように日本と台湾間で行き来するのか、どれくらいの頻度や規模で集団が混ざり合うのか、日本・台湾間の分布はどのようになっているか等の新たな疑問も生まれましたが、本研究はサクラエビの生態の理解に向けた大きな一歩といえます。現在、サクラエビの資源管理は日本・台湾で独自に行われていますが、本研究では両地域間の「つながり」が示され、今後はサクラエビの適切な資源管理や生態理解にむけて日本と台湾で協力関係が構築されることが期待されます。
発表者・研究者等情報
東京大学 大気海洋研究所
平井 惇也 講師
東海大学 海洋学部 海洋生物学科
西川 淳 教授
論文情報
雑誌名:Scientific Reports
題 名:Population panmixia of the pelagic shrimp Lucensosergia lucens between Japanese and Taiwanese waters in the western North Pacific
著者名:Junya Hirai*, Sheng-Tai Hsiao, Hsin-Ming Yeh, Jun Nishikawa*
DOI : 10.1038/s41598-025-91208-4
URL : https://doi.org/10.1038/s41598-025-91208-4![]()
研究助成
本研究は、環境省・(独)環境再生保全機構の環境研究総合推進費(JPMEERF20224R03)、科研費(23K26979)の支援により実施されました。本研究は「駿河湾海洋生態系研究プロジェクト(SUruga bay Research for Understanding Marine Ecosystems )」、通称SURUMEプロジェクトの一環として行われました。
用語解説
- (注1)ミトコンドリアDNA
- 細胞内小器官であるミトコンドリア内に存在するDNA。サクラエビでは約16000塩基対から成り、37遺伝子が存在する。母性遺伝する、塩基置換の起こる頻度が高いという特徴を有することから集団構造の把握にも活用されている。
- (注2)一塩基多型
- 集団の個体間のゲノム上で塩基配列が異なる場所を示し、SNPとも呼ばれる。一塩基多型の情報をゲノム上から広く集めることで、詳細な集団構造が把握可能になる。
- (注3)MIG-seq法
- 塩基配列情報が大量に決定可能な次世代シーケンサーを用い、ゲノム上の反復配列に挟まれた領域を効率的に取得し、これらの領域に含まれる一塩基多型を探索する手法。
- (注4)ゲノムスキミング
- 次世代シーケンサーを用いて断片化されたDNAの塩基配列を取得し、ミトコンドリアDNAの全長等の目的領域が復元可能な手法。全ゲノム配列の取得を目的としておらず、膨大な量の次世代シーケンサーのデータを必要としない。
問合せ先
東京大学 大気海洋研究所 海洋生命システム研究系 海洋生態系科学部門
講師 平井 惇也(ひらい じゅんや)
E-mail:hirai◎aori.u-tokyo.ac.jp ※「◎」は「@」に変換してください
![]()
![]()

 教職員募集
教職員募集 所内専用
所内専用
