窒素循環を駆動する微生物の網羅的検出法を開発 ―アンモニア酸化酵素遺伝子の選択的シークエンシングによる 遺伝的多様性解析―
2024年11月18日
東京大学
海洋研究開発機構

研究成果
発表のポイント
◆キャプチャーシークエンシング法に基づいた、窒素循環を駆動する微生物の網羅的検出法の開発に成功しました。
◆従来法に比べてバイアスが少なく、アンモニア酸化能を有する微生物群集を低バイアス・高効率・高解像度に検出することを実証しました。
◆本手法は、海洋をはじめ、さまざまな環境の微生物群集に応用可能です。

概要
東京大学大気海洋研究所の塩崎拓平准教授と伊知地稔特任研究員(研究当時)、海洋研究開発機構海洋機能利用部門の平岡聡史研究員らによる研究グループは、キャプチャーシークエンシング法(図1上段)を用いたアンモニア酸化(図2)を担う微生物の網羅的検出法を開発しました。模擬的な微生物群集サンプルを用いて、一般的に利用されるアンプリコンシークエンシング法(図1中段)とショットガンシークエンシング法(図1下段)と比較したところ、本手法は低バイアス・高効率にアンモニア酸化微生物の群集構造を復元できることが示されました。また、海洋サンプルに応用したところ、上述の一般手法と比較して、より高解像度にアンモニア酸化微生物群集を検出できることが示されました(図3)。本手法は海洋のみならず、土壌など他の環境サンプルを対象にすることも可能です。本手法の応用によって、地球上で窒素循環を駆動している微生物群集の多様性評価が進むと考えられます。
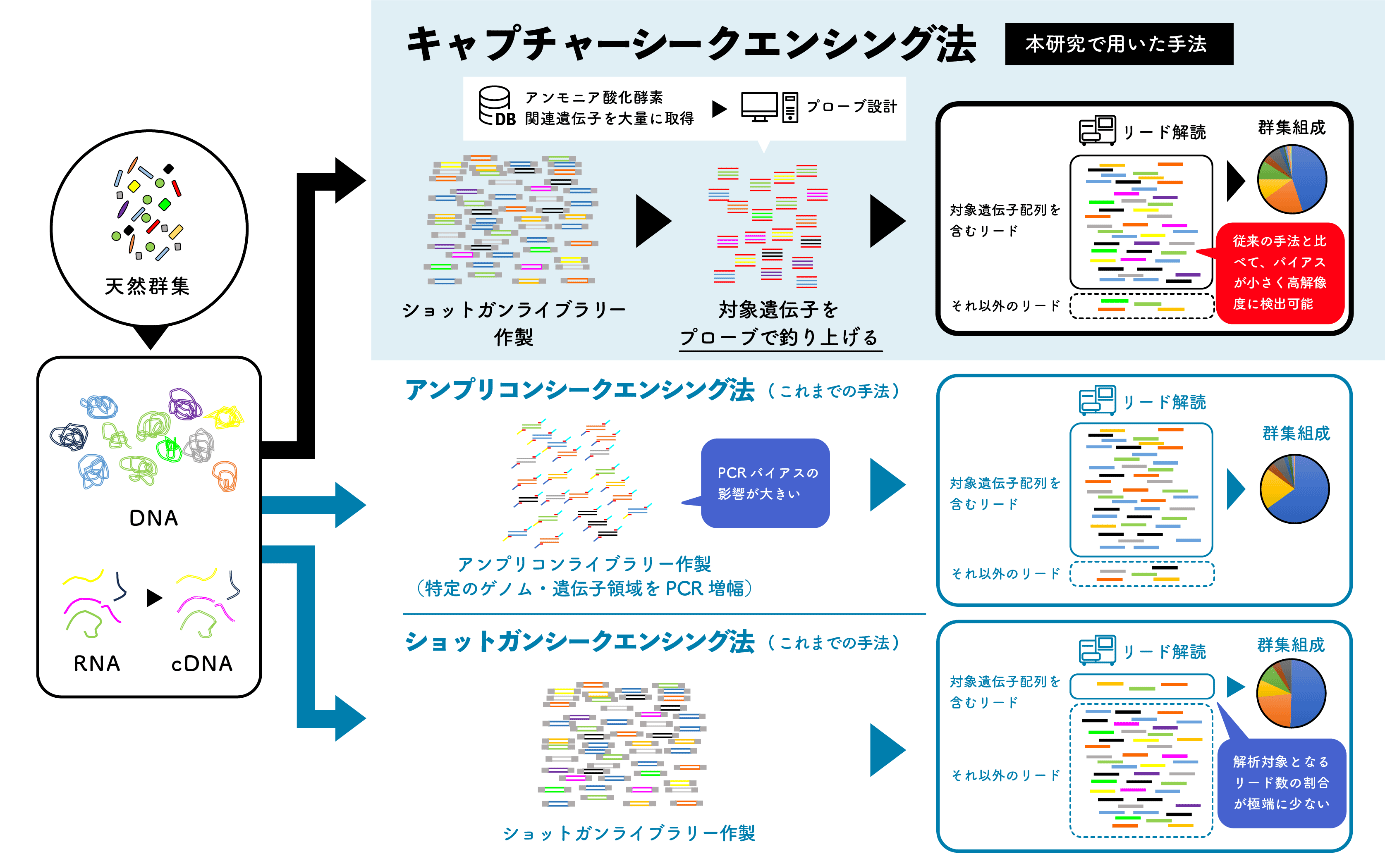
図1:環境中の微生物群集構造の解析手法(キャプチャーシークエンシング法・アンプリコンシークエンシング法・ショットガンシークエンシング法)の比較
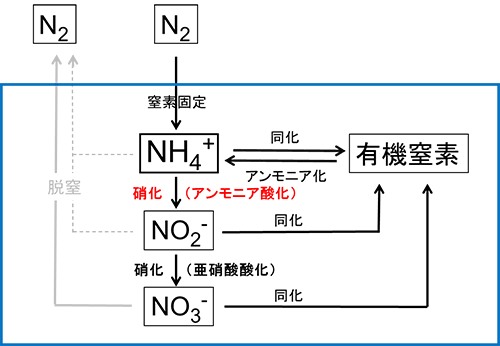
図2:自然界の微生物が駆動する窒素循環のモデル図
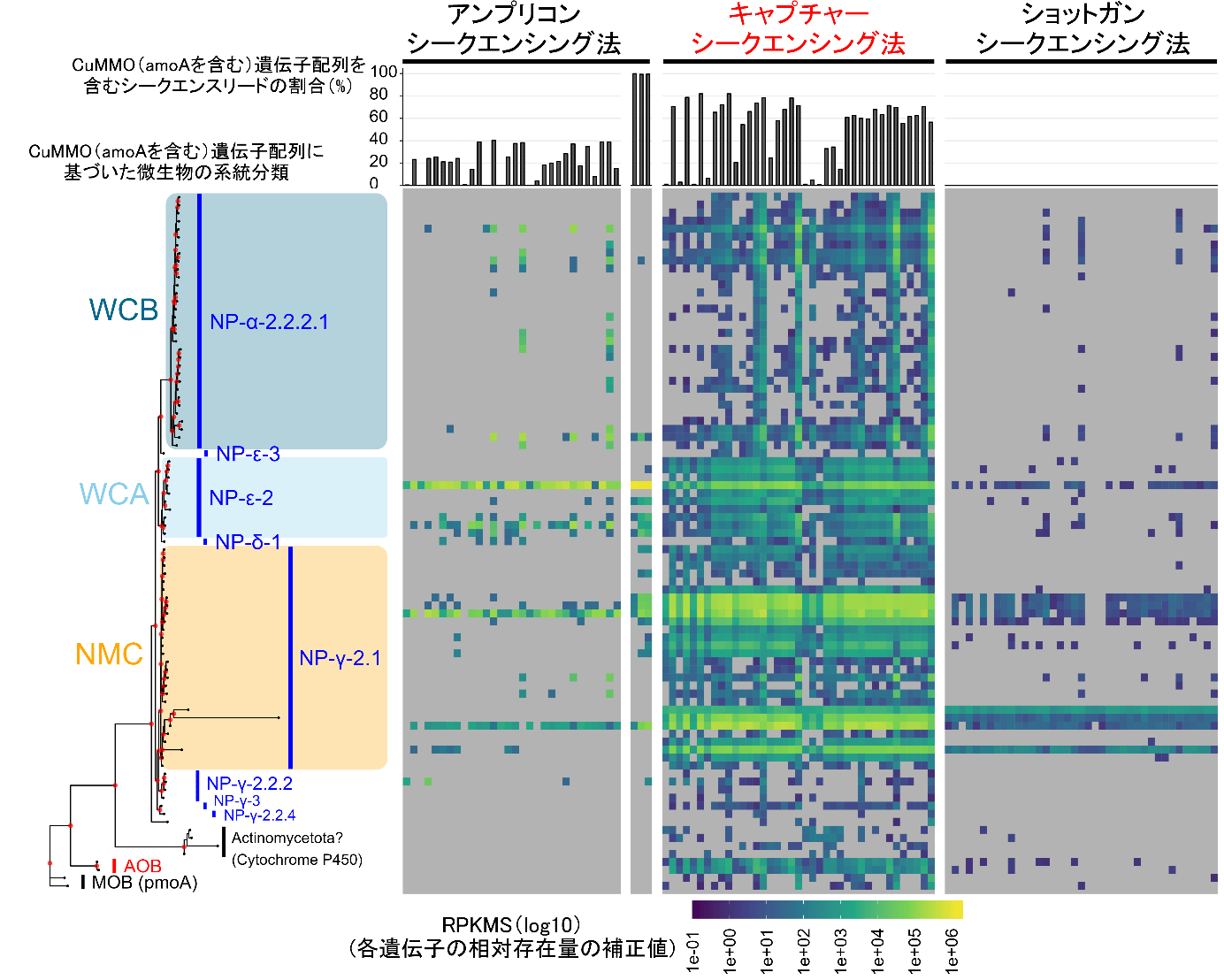
図3:海洋サンプルから検出されたamoA遺伝子配列の系統樹と、3手法から予測されたアンモニア酸化微生物の存在量の比較
キャプチャーシークエンシング法ではアンプリコンシークエンシング法やショットガンシークエンシング法と比較して、amoA遺伝子配列を含むシークエンスリードの割合が高く、解像度高くアンモニア酸化微生物の存在量予測ができている。
発表内容
微生物群集の網羅的検出には、アンプリコンシークエンシング法とショットガンシークエンシング法(いわゆるメタゲノムシークエンシング・メタトランスクリプトームシークエンシング)の2種類が広く利用されています(図1中・下段)。前者は種特異的な遺伝子やゲノム領域をターゲットに、プライマーと呼ばれる短いDNA断片(オリゴヌクレオチド)を用いてPCR増幅(注1)させ、そのPCR産物(アンプリコン)をシークエンシングすることで、遺伝的多様性から群集構造を推定する方法です。後者は、サンプル中に存在するDNA/RNAを網羅的にシークエンシングし、得られたリードに含まれる特定のターゲット配列だけを利用して群集構造を推定します。これらの手法は、近年の次世代シークエンサーの発展と普及に伴い、あらゆる環境中の微生物群集構造を調べるための最もスタンダードな手法になっています。しかしながら、両者は共に完璧な手法ではなく、いくつかの実験上不可避の問題が生じることも知られています。例えば前者は、プライマー配列に合致しない配列は増幅されないため多数の見落としが生じる、PCR増幅に様々なバイアスが生じるため得られる結果は元の群集構造を正確には反映できていない、後者は全リード中に占めるターゲット配列の割合が非常に小さくなるため、特に群集中の希少種は十分なデータが得られず多様性の正確な把握が困難である、といった問題です。
本研究ではこれらの問題を克服するために、第3の手法として、キャプチャーシークエンシング法に基づく方法論を開発しました(図1上段)。キャプチャーシークエンシング法は、主に医学研究分野で用いられている手法であり、例えばヒトゲノム中の疾患関連遺伝子の探索(エクソーム解析)などに利用されている技術です。本研究では、この技術を環境中の微生物群集解析に応用する試みを行いました。具体的には、アンモニア酸化を担う微生物系統群の検出と多様性評価を目的に、それら微生物が持つアンモニア酸化酵素遺伝子(amoA遺伝子)をターゲットとしました。
アンモニア酸化は、硝化と呼ばれる窒素循環プロセスの一部です。自然界では、動植物や微生物由来の有機物の一部は分解されてアンモニア(NH4+)となり、微生物の活動を介してさらに亜硝酸(NO2-)や硝酸(NO3-)といった無機窒素へと変換され、そのプロセスは硝化反応と呼ばれます。硝化反応は窒素循環の中心的な反応経路の一つであり、特に環境中の各無機窒素の割合を決定する点で重要です(図2)。特にアンモニア酸化の鍵酵素遺伝子(注2)であるamoA遺伝子は、アンモニア酸化微生物の系統推定にも利用可能なバイオマーカーとして、この遺伝子配列に基づいた研究が世界で進められています。今日、アンモニア酸化微生物を含む硝化微生物は、微生物全体から見るとごく一部に限られているものの、当初想定されていた以上に多くの種類が存在するなど、新知見が徐々に明らかになりつつあります。
しかしながら、方法論的限界もあり、アンモニア酸化微生物の多様性の理解は未だ十分ではありません。例えば、amoA遺伝子は微生物系統間で配列の多様性が高く、PCRでこの配列を増幅しようとしても、全アンモニア酸化微生物を網羅する「普遍的な」プライマーセットを設計することは本質的に困難です。そのためアンプリコンシークエンシング法を用いた解析では、アンモニア酸化能を持つ微生物のごく一部のみを解析対象とせざるを得ません。また、PCRバイアスも生じるために、正確な群集構造の推定が困難です。一方で、メタゲノム・メタトランスクリプトームシークエンシング法ではそのような制約は存在しないものの、アンモニア酸化微生物は環境中ではごく少数に過ぎないため、現実的なシークエンス量でその多様性を正確に捉えることに限界がありました。
本研究ではまず、公共遺伝子データベースからamoA遺伝子を含むスーパーファミリー(銅含有膜結合型モノオキシゲナーゼ; CuMMO)遺伝子を29878配列収集し、プローブを独自に設計しました。このプローブに対して、サンプル中のDNA断片を相補的に結合(ハイブリダイズ)させ、さらに磁力ビーズに結合(キャプチャ)させて精製することで、amoA遺伝子を含むDNA断片だけを選択的に濃縮することができます。次に、既知のamoA遺伝子を複数組み込んでデザインした模擬的な微生物群集サンプルを作成し、このキャプチャーシークエンシングを行う最適な実験条件を検討しました。さらに、この手法によって得られるデータの解析のため、独自のバイオインフォマティクス解析パイプラインを設計しました。
最初に、模擬的な微生物群集サンプルを利用して、本手法の妥当性を評価しました。この解析では比較として、一般的なアンプリコンシークエンシング法とメタゲノムシークエンシング法でも解析しました。その結果、アンプリコンシークエンシング法では特定の微生物系統由来の配列のみが選択的に検出される結果となり、メタゲノムシークエンシング法ではamoA遺伝子配列を含むリード数が少なすぎるために安定的な解析結果を得られませんでした。これらの結果は、現在一般的に利用されている2手法では実験技術上の重大なバイアスがあることを示しており、同手法を用いた今日までの研究結果にも疑問を投げかける結果です。一方で我々が提案するキャプチャーシークエンシング法は、高精度に群集構造を復元できることが示され、上述の2手法で生じるバイアスを克服できたことを示す結果でした。
次に、実際の環境サンプルとして海洋微生物サンプルを対象とした解析を実施しました(図3)。海洋研究開発機構(JAMSTEC)が運用する東北海洋生態系調査研究船「新青丸」を利用して東北沖海域の水深0~500mから海水を採取し、そこに存在する微生物群集が発現している遺伝子転写産物(メタトランスクリプトーム)をサンプルとして利用しました。解析の結果、キャプチャーシークエンシング法ではバクテリア・アーキア合わせて計80系統ものアンモニア酸化微生物を検出できましたが、そのうち44系統はアンプリコンシークエンシング法では検出できず、また36系統はショットガンシークエンシング法では検出できませんでした。本結果は、キャプチャーシークエンシング法を用いることで、従来は見逃されたり過小・過大評価されていた自然環境中のアンモニア酸化微生物系統を、より低バイアス・高効率・高解像度に評価できることを示した結果です。
窒素循環に関わる微生物をはじめ、地球上の様々な物質循環に関わる微生物は、群集中ではマイナーであることが多く、一見すると地味な存在に思われます。しかしながら、このような微生物は地球全体の生態系の土台を支え、根幹を維持する上で不可欠の存在であり、その微生物多様性を知ることは物質循環の頑強性や脆弱性を知ることに繋がります。本手法は、海洋以外のあらゆる環境微生物群集に対しても利用可能な、汎用的手法です。また、プローブ配列を設計し直すことで、アンモニア酸化以外の物質循環プロセスを駆動する微生物系統についても、同様に高効率・高精度な解析ができる可能性があります。地球温暖化で環境が変化しつつある現在において、本手法の改良と拡張、そして様々な自然環境サンプルを対象とした研究の展開が期待されます。
発表者・研究者等情報
東京大学大気海洋研究所
塩崎 拓平 准教授
伊知地 稔 研究当時:特任研究員
現:東京都立大学理学研究科 客員研究員
兼:株式会社キアゲンセールスアプリケーション部 スペシャリスト
海洋研究開発機構海洋機能利用部門
平岡 聡史 研究員
論文情報
雑誌名:Molecular Ecology Resources
題 名:Probe capture enrichment sequencing of amoA genes improves the detection of diverse ammonia-oxidizing archaeal and bacterial populations
著者名:Satoshi Hiraoka†*, Minoru Ijichi†, Hirohiko Takeshima, Yohei Kumagai, Ching-Chia Yang, Yoko Makabe-Kobayashi, Hideki Fukuda, Susumu Yoshizawa, Wataru Iwasaki, Kazuhiro Kogure, Takuhei Shiozaki*
† 共同主著者、* 共同責任著者
DOI:10.1111/1755-0998.14042
URL:https://doi.org/10.1111/1755-0998.14042![]()
研究助成
本研究は、文部科学省海洋資源利用促進技術開発プログラム「海洋生物ビッグデータ活用技術高度化」(課題番号:JPMXD1521474594)、科研費基盤研究(B)(課題番号:JP19H04263)、若手研究(課題番号:JP20K15444)、基盤研究(B)(課題番号:JP21H03592)、基盤研究(B)(課題番号:JP22H03716)、基盤研究(B)(課題番号:JP23H05411)、学術変革領域研究(学術研究支援基盤形成)(課題番号:JP22H04925)、JST CREST「海洋生物多様性および生態系の保全・再生に資する基盤技術の創出」(課題番号:JPMJCR11A3)、「ゲノムスケールのDNA設計・合成による細胞制御技術の創出」(課題番号:JPMJCR19S2)の支援により実施されました。
用語解説
- (注1)PCR
- Polymerase Chain Reactionの略で、日本語では「ポリメラーゼ連鎖反応」と呼ばれる、特定のゲノムや遺伝子領域のDNAを増幅する手法です。
- (注2)鍵酵素遺伝子
- 代謝経路で反応速度の最も速い段階を触媒する酵素の遺伝子を指します。
問合せ先
東京大学大気海洋研究所
准教授 塩崎 拓平(しおざき たくへい)
E-mail:shiozaki◎g.ecc.u-tokyo.ac.jp
海洋研究開発機構海洋機能利用部門
研究員 平岡 聡史 (ひらおか さとし)
E-mail:hiraokas◎jamstec.go.jp
※「◎」は「@」に変換してください
![]()
![]()

 教職員募集
教職員募集 所内専用
所内専用
