メタゲノムとエピゲノムを融合した「メタエピゲノム」解析の提唱と実証 ~環境細菌叢が持つゲノム修飾機構の広大な未開拓領域の解明へ~
2019年1月11日
東京大学 大学院理学系研究科
東京大学 大学院新領域創成科学研究科
東京大学 大気海洋研究所
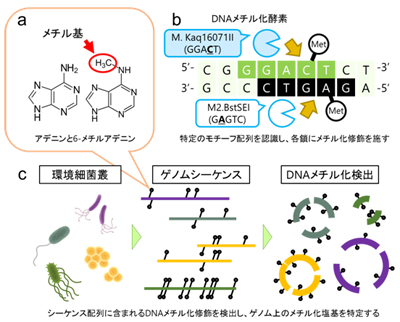
近年、細胞の癌化や分化を制御する機構としてエピジェネティクスが注目を集めており、哺乳類ではヒトやマウス等をモデルにエピゲノム解析が盛んに行われています。一方で、細菌・古細菌といった原核生物においてもこのエピジェネティクスという現象が起きていることが知られており、さまざまな分離培養株を用いた研究が古くから進められてきました。しかしながら、環境中の原核生物の大半は培養が難しく、原核生物の主なエピジェネティクス機構であるDNAメチル化修飾の実験的観測が困難なために、環境細菌叢におけるDNAメチル化修飾の普遍性や多様性は全く不明でした。本研究において、東京大学の平岡聡史大学院生(現:海洋研究開発機構 特任研究員)、按田瑞恵特別研究員、岩崎渉准教授と、京都大学、遺伝学研究所との共同研究チームは、第3世代シーケンサーと呼ばれる1分子DNAシーケンサーを活用することで、環境細菌叢のDNAメチル化修飾を観測する新たな手法「メタエピゲノム解析」を提唱し、その有効性を実証しました。本手法を用いて滋賀県の琵琶湖に生息する淡水細菌叢を解析したところ、多様なDNAメチル化モチーフ配列を検出することに成功し、さらに驚くべきことに、その約半数は新規のモチーフ配列であることが分かりました。加えて、バイオインフォマティクスによるDNAメチル化酵素遺伝子の予測と大腸菌を用いた遺伝子組み換え実験を行い、それらのモチーフ配列を特異的に認識する新規のDNAメチル化酵素を複数発見しました。原核生物は海洋、土壌、腸内など、地球上のあらゆる環境に存在しています。今後、メタゲノム解析に加えてメタエピゲノム解析を進めていくことで、原核生物のエピジェネティクスが駆動する生理・生態メカニズムの解明に繋がっていくことが期待されます。
詳しくはこちらをご覧下さい。
東京大学大学院理学系研究科プレスリリース![]() (2019年1月16日)
(2019年1月16日)
![]()

 教職員募集
教職員募集 所内専用
所内専用
